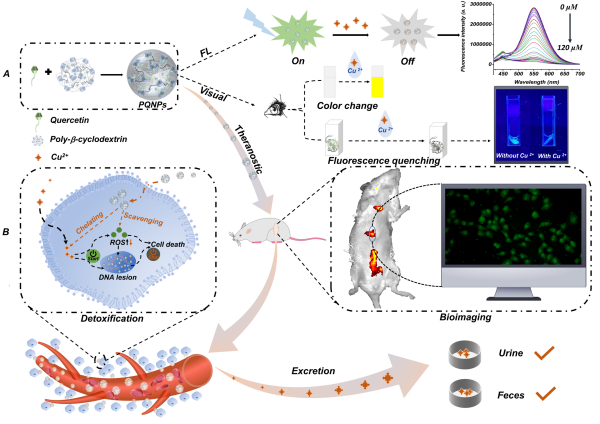
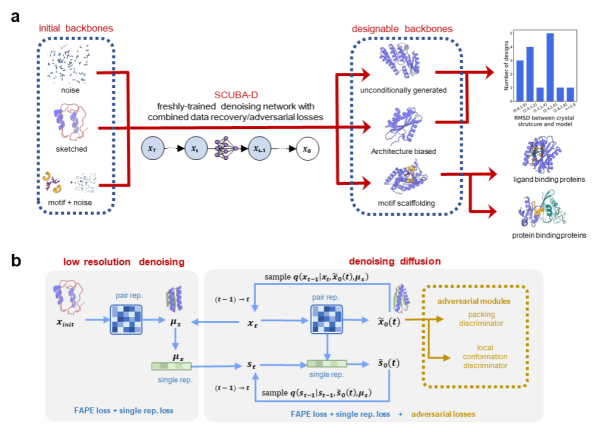
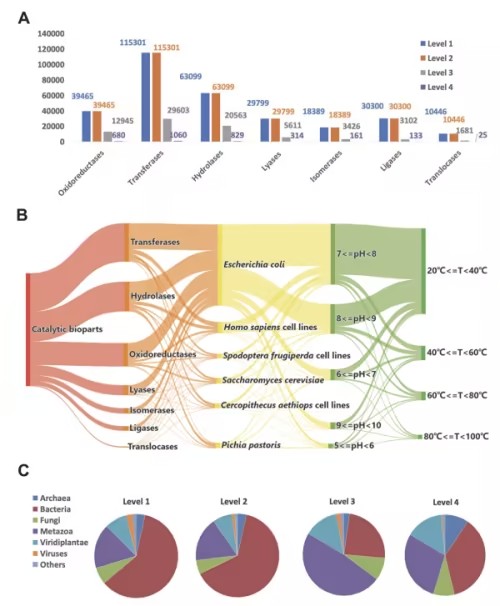
近日,中国科学院上海营养与健康研究所研究员张国庆与分子植物科学卓越创新中心的周志华研究组联合发布了题为《RDBSB: a database for
catalytic bioparts with experimental
evidence》的研究论文,介绍了一个全新的催化元件数据库RDBSB。该数据库旨在为合成生物学设计提供实验验证的催化元件,并构建了一个在线查询平台(https://www.biosino.org/rdbsb),同时还研发了名为PathFinder的工具,以便用户根据底物和目标产物设计相应的合成途径。 催化元件在设计、构建和优化代谢途径以及生命系统中起着基础性作用。为了满足合成生物学的需求,构建一个可靠且经过实验验证的催化元件数据库显得尤为重要。然而,现有的生物元件库往往缺乏诸如底盘信息、催化反应的最佳pH和温度等关键信息,限制了合成过程的优化。因此,研究团队整合了390,708个催化元件,通过审编后得到83,193个经过实验验证的催化元件,这些元件中包含大量转移酶、水解酶和氧化还原酶。 RDBSB的设计考虑了催化元件的活性、底物、最佳反应条件等多个关键参数,为科研人员提供了丰富的信息资源。特别值得一提的是,该数据库中有3200个催化元件详细列出了其最适催化反应温度和最适pH值等重要信息,这些元件大多来源于细菌、后生动物和植物,展示了不同生物体中催化反应的多样性。 研究团队的努力不仅体现在数据的整合上,还在于在线工具的开发。RDBSB提供了包括MapView、AlphaFold、PVQD和PathFinder等在内的多种在线元件和途径设计工具。PathFinder工具特别引人注目,它能够帮助用户设计出如淫羊藿素和棉铃虫性信息素等复杂化合物的人工合成途径。通过这个工具,研究人员可以完整复现相关合成过程,提高了实验设计的效率和可行性。 用户在RDBSB上可以在线搜索催化元件的详细信息,并查阅相关文献。此外,平台还支持用户提交新的催化元件,实现数据的快速共享与利用。至今,RDBSB已为一千多个催化元件的共享提供了支撑,这无疑将极大丰富合成生物学途径设计的资源库。 这一研究工作得到了国家重点研发计划和中国科学院战略生物资源服务网络计划等的支持。随着RDBSB数据库的推出,科研人员将获得必要的工具,以便在合成生物学领域开展更深入的研究。未来,RDBSB将为生物工程、医药开发等领域提供更加可靠的实验数据支持,推动合成生物学的进步与应用。 综上所述,RDBSB的构建不仅为催化元件的研究提供了重要的数据库支持,还为合成生物学领域的科研人员提供了创新的工具和平台,助力其在催化途径设计上的探索与实现。随着更多数据的积累和工具的完善,RDBSB有望在未来的科研工作中发挥越来越重要的作用。